Pheatmap in R: 사용자 정의 가능한 클러스터 히트맵 생성
Published on
히트맵은 데이터 과학자의 도구 상자에서 필수적인 도구로서 복잡한 데이터 세트의 시각적 직관성을 제공합니다. R에서 사용 가능한 다양한 패키지 중에서도 Pheatmap은 그 유연성과 사용자 정의 옵션으로 두드러집니다. 이 문서에서는 R의 Pheatmap을 사용하여 아름다우면서도 사용자 정의 가능한 클러스터 히트맵을 생성하는 과정을 안내합니다.
Pheatmap은 단순히 R의 기능이 아닌, 기존 R heatmap 함수보다 훨씬 더 많은 제어와 사용자 정의 옵션을 제공하는 강력한 도구입니다. Pheatmap을 사용하면 사용자는 유전자 발현 분석을 시각화하고, 상관 관계 히트맵을 그리며, 레이블 크기와 덴드로그램 가시성을 사용자 정의할 수 있습니다. Pheatmap의 세계로 빠져들어 그 기능을 탐색해보겠습니다.
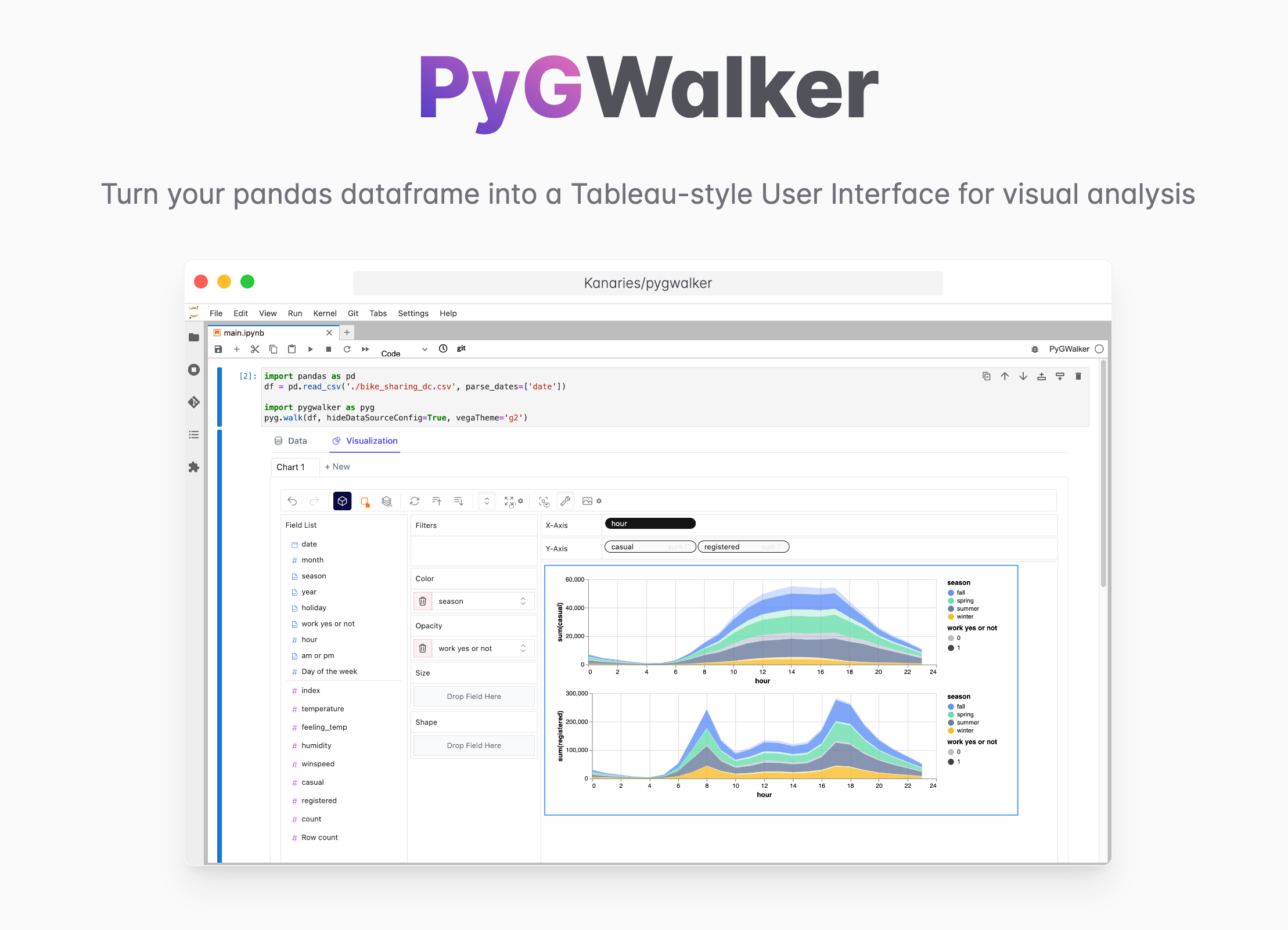
파이썬 판다스 데이터프레임에서 코드 없이 빠르게 데이터 시각화를 생성하고 싶으신가요?
PyGWalker는 시각화를 통한 탐색적 데이터 분석을 위한 파이썬 라이브러리입니다. PyGWalker (opens in a new tab)는 파이썬, 주피터 노트북 환경에서 사용 가능하며, 판다스 데이터프레임과 폴라 데이터프레임을 Tableau 스타일의 사용자 인터페이스로 변환하여 시각적 탐색을 단순화합니다.
R의 Pheatmap이란?
Pheatmap은 R에서 복잡한 데이터를 단순화하여 시각화하는 아름다운 히트맵을 생성하는 기능입니다. 기본 R 히트맵 함수인 heatmap() 및 heatmap.2()와 비교하여 더 많은 제어 및 사용자 정의 옵션을 제공합니다. Pheatmap은 예쁘고 정보적인 히트맵을 생성할 수 있는 능력으로 뛰어납니다.
Pheatmap은 유전체학에서 특히 유전자 발현 데이터를 시각화하는 데에 유용하게 사용됩니다. 주석을 추가하고 유사한 데이터를 그룹화하는 군집화 방법을 사용하여 히트맵의 해석 가능성을 향상시킵니다. 또한 행/열 Z-점수 표준화 옵션을 제공하여 특정 데이터 분석 시나리오에서 중요할 수 있습니다.
Pheatmap은 어떻게 작동하나요?
Pheatmap은 데이터 행렬을 가져와 시각적으로 직관적인 히트맵으로 변환하는 방식으로 작동합니다. 데이터 값을 히트맵에서 색상으로 표현하며, 색상의 강도는 값을 나타냅니다. 이를 통해 데이터의 패턴과 상관 관계를 쉽게 식별할 수 있습니다.
이 함수는 데이터에 계층적 군집화를 수행하여 유사한 행과 열을 함께 그룹화합니다. 이는 데이터 포인트 간의 계층적 관계를 나타내는 트리 모양의 다이어그램인 덴드로그램으로 시각적으로 표시됩니다. Pheatmap이 사용하는 군집화 방법은 사용자의 요구에 따라 사용자 정의할 수 있습니다.
Pheatmap은 또한 히트맵의 외관을 매우 많이 사용자 정의할 수 있습니다. 사용자는 색 팔레트, 레이블 크기, 덴드로그램 가시성 등을 제어할 수 있습니다. 이를 통해 Pheatmap은 R에서 데이터 시각화를 위한 다재다능한 도구로 사용할 수 있습니다.
표준 R heatmap에 비해 Pheatmap의 장점
기본 R 히트맵 함수는 기본적인 히트맵 생성에 유용하지만, Pheatmap은 많은 데이터 과학자들에게 선호되는 몇 가지 장점을 제공합니다.
첫째로, Pheatmap은 히트맵의 외관에 대해 더 많은 제어를 제공합니다. 사용자는 색 팔레트를 사용자 정의하고, 레이블 크기를 조정하며, 덴드로그램의 가시성을 제어할 수 있습니다. 이를 통해 정보 전달력뿐만 아니라 시각적인 매력까지 갖춘 히트맵을 생성할 수 있습니다.
둘째로, Pheatmap은 데이터에 계층적 군집화를 수행하여 비슷한 행과 열을 함께 그룹화합니다. 이는 히트맵의 해석 가능성을 향상시키고 데이터의 패턴을 쉽게 식별할 수 있도록 합니다.
셋째로, Pheatmap은 주석 추가 및 필터 사용을 가능하게 합니다. 이는 유전자 발현 분석에서 특히 유용할 수 있습니다. 행/열 Z-점수 표준화 옵션도 제공하여 데이터 분석의 유연성을 더욱 확보할 수 있습니다.
마지막으로, 기본 R 히트맵 함수는 기본적인 히트맵 생성에 유용한 도구이지만, Pheatmap은 더 많은 제어와 사용자 정의를 제공하여 R에서 데이터 시각화에 강력한 도구로 사용할 수 있습니다.
R에서 Pheatmap 외관 사용자 정의하기
Pheatmap의 주요 장점 중 하나는 특정 요구에 맞게 히트맵의 외관을 사용자 정의할 수 있다는 점입니다. 다음은 사용자 정의하는 방법입니다:
색상 사용자 정의
Pheatmap은 히트맵에서 사용되는 색 팔레트를 사용자 정의할 수 있습니다. 이는 pheatmap() 함수의 color 매개변수를 사용하여 수행할 수 있습니다. 기존에 R에서 제공하는 다양한 색 팔레트 중에서 선택하거나 자체적으로 생성할 수 있습니다.
레이블 사용자 정의
히트맵에서 레이블의 크기와 외관은 fontsize 및 fontface 매개변수를 사용하여 조정할 수 있습니다. 이를 통해 히트맵의 가독성을 제어하고 프레젠테이션에 맞게 조정할 수 있습니다.
덴드로그램 가시성
Pheatmap은 데이터 포인트 간의 계층적 관계를 보여주는 트리 모양 다이어그램인 덴드로그램의 가시성을 제어할 수 있습니다. 이는 pheatmap() 함수의 show_rownames 및 show_colnames 매개변수를 사용하여 수행할 수 있습니다.
주석 추가
Pheatmap은 히트맵에 주석을 추가할 수 있습니다. 이는 유전자 발현 분석에서 특히 유용할 수 있습니다. 이는 pheatmap() 함수의 annotation_row 및 annotation_col 매개변수를 사용하여 수행할 수 있습니다.
Pheatmap를 사용하면 정보성과 시각적으로 매력적인 열지도를 생성할 수 있는 고도로 맞춤화된 기능을 제공합니다. 유전체 데이터를 시각화하거나 상관관계 열지도를 그리는 경우에도 Pheatmap은 R에서 아름답고 맞춤화된 클러스터링 열지도를 생성할 수 있는 유연성과 제어력을 제공합니다.
Pheatmap에서 사용하는 클러스터링 방법
Pheatmap은 유사한 데이터 포인트를 그룹화하는 계층적 클러스터링 방법을 사용합니다. 이는 클러스터의 계층 구조를 구축하려는 클러스터 분석 방법입니다. 최종 결과는 데이터의 트리 기반 표현인 덴드로그램입니다. 이를 통해 사용자는 데이터 포인트 간의 관계를 강조하여 데이터를 시각화할 수 있습니다.
Pheatmap에서 클러스터링 방법은 각각 행과 열에 대해 clustering_distance_rows 및 clustering_distance_cols 매개변수를 사용하여 맞춤 설정할 수 있습니다. 기본 방법은 "유클리드"이지만 "최대", "맨해튼", "캔버라", "이진" 또는 "민코프스키"와 같은 다른 방법도 사용할 수 있습니다.
Pheatmap을 사용하여 R에서 열지도 그리기
R에서 Pheatmap을 사용하여 열지도를 생성하는 것은 간단합니다. 다음은 기본적인 예시입니다:
# pheatmap 라이브러리 로드
library(pheatmap)
# 데이터 행렬 생성
data <- matrix(rnorm(200), 20, 10)
# 열지도 생성
pheatmap(data)기본 설정으로 기본 열지도가 생성됩니다. pheatmap() 함수에 매개변수를 추가하여 열지도를 사용자 정의할 수 있습니다. 예를 들어, 색상 팔레트를 변경하려면 color 매개변수를 사용할 수 있습니다:
# 색상 팔레트 정의
my_palette <- colorRampPalette(c("blue", "white", "red"))(25)
# 사용자 정의 색상 팔레트로 열지도 생성
pheatmap(data, color = my_palette)Pheatmap에서 색상 맞춤 설정하기
Pheatmap은 매우 높은 정도의 색상 맞춤 설정을 지원합니다. 사용자 정의 색상 팔레트를 정의하고 열지도에 적용할 수 있습니다. 이를 위해 pheatmap() 함수에서 color 매개변수를 사용합니다. 다음은 예시입니다:
# 색상 팔레트 정의
my_palette <- colorRampPalette(c("blue", "white", "red"))(25)
# 사용자 정의 색상 팔레트로 열지도 생성
pheatmap(data, color = my_palette)이 예시에서 colorRampPalette() 함수를 사용하여 파란색에서 흰색에서 빨간색까지 25개의 색상으로 구성된 팔레트를 생성합니다. 그런 다음 color 매개변수를 사용하여 열지도에 이 팔레트를 적용합니다.
결론
결론적으로, Pheatmap은 R에서 맞춤화된 클러스터링 열지도를 생성하는 강력한 도구입니다. 유전체 데이터를 시각화하거나 상관관계 열지도를 그리거나 데이터를 탐색하는 경우에도 Pheatmap은 필요한 유연성과 제어력을 제공합니다.
자주 묻는 질문
표준 기본 R 열지도에 비해 Pheatmap을 사용하는 장점은 무엇인가요?
Pheatmap은 표준 기본 R 열지도 함수보다 여러 가지 이점을 제공합니다. 열지도의 외형을 더욱 제어할 수 있으며 데이터에 계층적 클러스터링을 수행하고 주석을 추가하고 필터를 사용할 수 있습니다. 이는 R에서 데이터 시각화를 위한 강력한 도구로 만듭니다.
Pheatmap에서 색상 팔레트를 어떻게 맞춤 설정할 수 있나요?
Pheatmap에서는 pheatmap() 함수의 color 매개변수를 사용하여 색상 팔레트를 맞춤 설정할 수 있습니다. R에서 제공하는 다양한 색상 팔레트 중 선택하거나 직접 만들 수 있습니다.
Pheatmap은 어떤 클러스터링 방법을 사용하나요?
Pheatmap은 유사한 데이터 포인트를 그룹화하기 위해 계층적 클러스터링을 사용합니다. 클러스터링 방법은 clustering_distance_rows 및 clustering_distance_cols 매개변수를 사용하여 맞춤 설정할 수 있습니다. 기본 방법은 "유클리드"이지만 "최대", "맨해튼", "캔버라", "이진" 또는 "민코프스키"와 같은 다른 방법도 사용할 수 있습니다.